✓ Traitement automatique des commandes en ligne 24 h/24 7 j/7
✓ Assistance technique et produits pertinente et professionnelle
✓ Commande (ou réapprovisionnement) rapide et fiable
therascreen NRAS Pyro Kit (24)
Cat. No. / ID: 971530
✓ Traitement automatique des commandes en ligne 24 h/24 7 j/7
✓ Assistance technique et produits pertinente et professionnelle
✓ Commande (ou réapprovisionnement) rapide et fiable
Caractéristiques
- Conforme à la directive européenne IVD 98/79/CE
- Résultats complets en temps-réel
- Quantification précise de mutations du gène NRAS
- Interprétation aisée des informations de séquences complexes
Détails produit
Performances
Linéarité
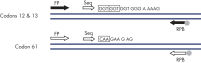
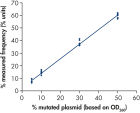
La linéarité a été déterminée à l’aide de mélanges de plasmides porteurs de la séquence sauvage ou mutante pour les mutations GGT -> GAT au niveau des codons 12 et 13 et la mutation CAA -> CGA au niveau du codon 61 (voir figure « Linéarité de la mutation GGT -> GAT »). Les plasmides ont été mélangés dans des proportions permettant d’obtenir quatre niveaux de mutation (5, 10, 30 et 50 %). Chaque mélange a été analysé avec trois lots différents du kit therascreen NRAS Pyro, lors de trois analyses de pyroséquençage portant chacune sur trois réplicats. (1)
Les résultats étaient linéaires, avec une non-linéarité autorisée de 5 unités % dans l’intervalle testé de 5 à 50 % de niveau de mutation. Des résultats similaires ont été obtenus pour les mutations GGT -> GAT au niveau du codon 13 et CAA -> CGA au niveau du codon 61. (1)
Précision
Les données de précision permettent de déterminer la variabilité totale des tests. Elles ont été obtenues pour trois niveaux différents, par analyse des mélanges de plasmides susmentionnés, avec trois réplicats chacun. (1)
La répétabilité (variabilité intra-test et inter-lot) a été calculée à partir des données utilisées pour déterminer la linéarité (trois analyses réalisées le même jour avec divers lots du kit therascreen NRAS Pyro). La précision moyenne (variabilité intra-laboratoire) a été déterminée lors de trois analyses réalisées dans un seul laboratoire, trois jours différents, par des opérateurs, sur des systèmes PyroMark Q24 et avec des lots du kit therascreen NRAS Pyro variables. La reproductibilité (variabilité inter-laboratoire) a été calculée à partir de deux analyses réalisées chacune dans un laboratoire interne et dans un laboratoire externe, avec divers lots du kit therascreen NRAS Pyro. (1)
La répétabilité, la précision moyenne et la reproductibilité de la mutation GGT -> GAT au niveau du codon 12 étaient respectivement de 1,2–1,9, 1,0–2,0 et 1,3–3,1 unités de %, dans les limites mesurées d’un niveau de mutation compris entre 5 et 50 %. Des résultats similaires ont été obtenus pour les mutations GGT -> GAT au niveau du codon 13 et CAA -> CGA au niveau du codon 61. (1)
| % de plasmides mutés | Répétabilité (moyenne, É.T.) | Précision moyenne (moyenne, É.T.) | Reproductibilité (moyenne, É.T.) |
|---|---|---|---|
| 5 | 7,5 ; 1,2 | 7,3 ; 1,0 | 6,7 ; 1,3 |
| 10 | 14,6 ; 1,3 | 13,5 ; 1,1 | 13,7 ; 1,3 |
| 30 | 37,8 ; 1,9 | 37,9 ; 1,5 | 36,1 ; 2,9 |
| 50 | 59,8 ; 1,7 | 60,4 ; 2,0 | 57,5 ;, 3,1 |
Principe
Le kit therascreen NRAS Pyro est utilisé pour les mesures quantitatives de mutations des codons 12, 13 et 61 du gène NRAS humain en temps réel, à l’aide de la technologie de pyroséquençage sur le système PyroMark Q24. Le gène NRAS encode un homologue de l’oncogène viral du neuroblastome RAS de la GTPase (v-ras). Les mutations de NRAS sont présentes dans environ 13 à 25 % de l’ensemble des mélanomes malins et interviennent fréquemment dans les codons 12, 13 et 61. Ces mutations entraînent l’activation constitutive de voies de signalisation du NRAS. (1)
Les mutations suivantes sont détectées :
- Codon 12/13 : G12A, G12C, G12D, G12R, G12S, G12V, G13A, G13C, G13D, G13R, G13S, G13V
- Codon 61 : Q61E, Q61H, Q61K, Q61L, Q61P, Q61R
Procédure
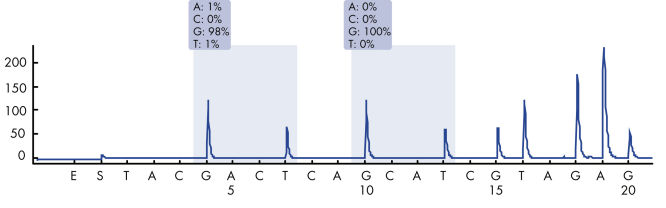
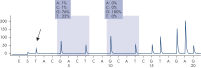
Après l’utilisation des amorces PCR ciblant les codons 12/13 et le codon 61, les amplicons sont immobilisés sur des billes de sépharose recouvertes de streptavidine (Streptavidin Sepharose High Performance). L’ADN simple brin est préparé et les amorces de séquence correspondantes s’hybrident avec l’ADN. Les échantillons sont ensuite analysés sur le système PyroMark Q24 à l’aide d’un fichier de configuration d’analyse et d’un fichier d’analyse. La fonction « Sequence to Analyze » (séquence à analyser) peut être ajustée afin de détecter les mutations rares après l’analyse (voir figures « Tracé de pyrogramme de la mutation GGT -> AGT au niveau de la base 1 du codon 12 » et « Tracé de pyrogramme après la réanalyse de l’échantillon dans la figure précédente »). (1)
Applications
Le kit therascreen NRAS Pyro est utilisé pour les mesures quantitatives de mutations des codons 12,13 et 61 du gène NRAS humain. Ce kit a été conçu pour faciliter l’identification des patients atteints de cancer les plus à même de bénéficier de traitements agissant spécifiquement sur le gène NRAS ou sur les sites dans lesquels le gène NRAS joue un rôle déterminant.
Les mutations suivantes sont détectées :
- Codon 12 : G12A, G12C, G12D, G12R, G12S, G12V
- Codon 13 : G13A, G13C, G13D, G13R, G13S, G13V
- Codon 61 : Q61E, Q61H, Q61K, Q61L, Q61P, Q61R
Données et illustrations utiles
Pyrogram trace of a normal genotype in codon 12-13.